Cell Stem Cell | 高绍荣教授团队与广东省第二人民医院欧湘红合作揭示人类早期胚胎发育过程中时期特异性组蛋白H3K9me3修饰调控逆转座子沉默的机制
组蛋白H3K9me3修饰,作为异染色质的标志之一,对哺乳动物的胚胎发育和细胞命运决定具有十分重要的作用[1, 2]。H3K9me3的不完全去除是体细胞克隆胚胎发育阻滞在合子基因组激活(zygotic genome activation, ZGA)时期的关键原因[3, 4],而富集H3K9me3的异染色质的过早建立也会严重阻碍小鼠着床前胚胎发育[5]。在人类和小鼠的ZGA阶段(人类主要在8细胞期,小鼠主要在2细胞期),大量转座子被活跃转录,其中就包括长末端重复序列元件(long terminal repeats, LTR)成员MERVL/HERVL等[6]。MERVL/HERVL的时期特异性激活依赖于转录因子DUX,驱使胚胎进入全能性的状态[7-9]。由于LTR的转座和自我复制能力对哺乳动物基因组的完整性带来潜在的危害,其表达必须受到严格的调控。高绍荣课题组的前期研究发现在小鼠早期胚胎发育过程中存在由DNA甲基化向组蛋白H3K9me3修饰的转换以实现对LTR的沉默调控[10]。此外,也有研究强调在谱系特异性基因启动子区域的H3K9me3修饰对于指导后期细胞命运决定和谱系分化具有重大意义[2, 10]。然而,受到组蛋白修饰检测技术和人类着床前胚胎材料稀缺的限制,人们对H3K9me3修饰在人早期胚胎发育过程中的重编程机制及功能并不十分清楚。

2022年6月24日,3344体育官方入口高绍荣/刘晓雨/王晨飞团队与广东省第二人民医院欧湘红团队合作在Cell Stem Cell杂志上在线发表题为Stage-specific H3K9me3 occupancy ensures retrotransposon silencing in human preimplantation embryos的研究论文。该研究利用CUT&RUN技术[11],描绘了人类着床前胚胎发育过程中组蛋白H3K9me3修饰的动态变化图谱,并揭示了时期特异性H3K9me3修饰在逆转座子上的建立及其功能,发现了可能介导H3K9me3修饰建立的关键调控因子。

图1 人类着床前胚胎样品收集示意图
为了探究H3K9me3修饰在人类着床前胚胎发育过程中的动态变化特点,研究人员收集了人MII期卵母细胞、4-细胞、8-细胞和囊胚期的胚胎,并利用CUT&RUN技术对各时期H3K9me3、H3K4me3和H3K27me3修饰水平进行了检测(图1)。与预期相符的是,H3K9me3修饰在人早期胚胎中也是高度富集在LTR区域,并且富集H3K9me3的异染色质是逐渐建立起来的(图2)。此外,与小鼠相比,人早期胚胎中存在着相对保守的由H3K9me3介导的LTR沉默调控机制。
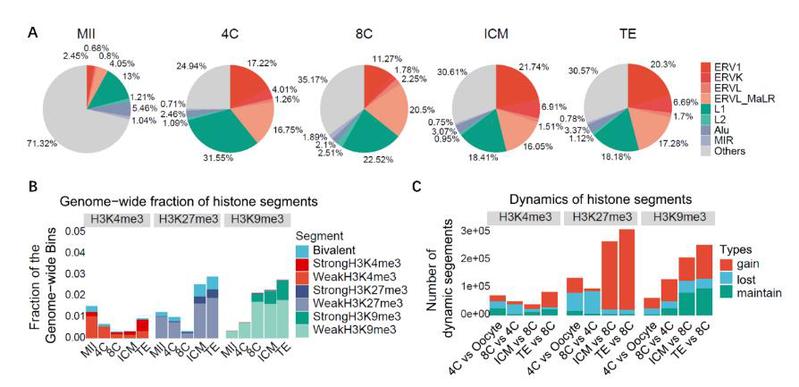
图2 H3K9me3修饰在基因组上的分布及其动态变化
研究人员发现,在人的早期胚胎中H3K9me3修饰在LTR区域上的建立是有时期特异性的,这确保了在不同胚胎阶段表达的LTR家族的有序沉默。有趣的是,分析结果显示,在人8-细胞中临时建立的H3K9me3修饰在随后的发育阶段更容易富集H3K4me1和H3K27ac修饰,表明其修饰区域在后期可能转变为增强子,而在囊胚中建立的H3K9me3修饰则更加稳定(图3)。
为了研究时期特异性H3K9me3修饰的建立是如何调控的,研究人员根据转录因子的结合位点以及H3K9me3修饰的分布情况预测了可能参与这一调控的转录因子,结果表明,一些经典的与H3K9me3修饰相关的调控因子(包括TRIM28和KRAB-ZNFs)可能参与调控囊胚特异的H3K9me3标记的LTR,而激活型转录因子DUX4和其他KRAB-ZNFs则可能参与调控8-细胞特异的H3K9me3标记的LTR(图3)。由于无法直接在人类胚胎中进行这些调控因子的功能验证,而分析发现小鼠早期胚胎发育过程中恰好也存在着与人类高度保守的时期特异性的H3K9me3修饰的调控,研究人员选择了这些候选因子(DUX4—8细胞特异性和ZNF808—囊胚特异性)在小鼠中的同源物(Dux和Zfp51)进行分析。借助课题组在之前的工作中构建的Dux基因敲除小鼠模型,研究人员发现Dux的缺失会同时影响卵裂期特异性(对应于人8-细胞特异性)和囊胚期特异性H3K9me3修饰的建立,并且可能同时存在依赖或不依赖于Zfp的两种调控LTR表达的方式;另一方面,在小鼠受精卵中进行siRNA注射从而敲降胚胎中的Zfp51后,研究人员发现缺乏Zfp51显著影响了囊胚特异性LTR上H3K9me3的建立,而其他LTR则不受影响。以上结果表明,Dux和Zfp51均可以调控小鼠早期胚胎LTR区域时期特异性H3K9me3的建立,并且精准调控LTR的沉默对正常胚胎发育具有重要意义。
最后,该研究还揭示了H3K4me3/H3K9me3二价结构域在人类植入前胚胎发育过程中是逐步建立的,可能与H3K4me3/H3K27me3二价结构域类似,起到调节谱系分化的作用。
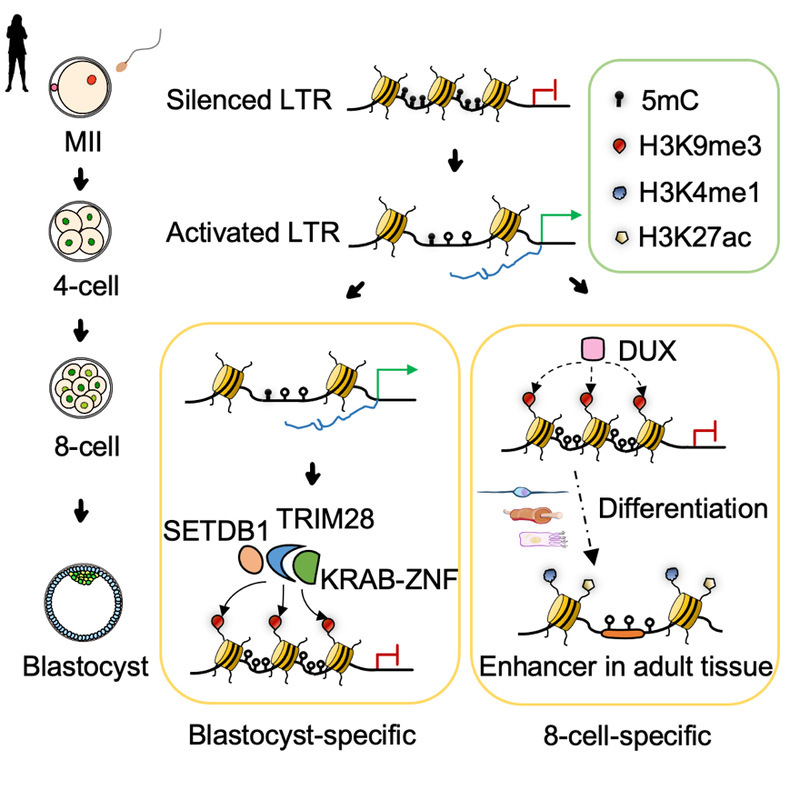
图3 人类早期胚胎发育过程中H3K9me3标记的LTR调控机制模式图
综上所述,该研究揭示了人类胚胎ZGA和第一次谱系分化过程中的H3K9me3修饰重编程过程,描绘了人类植入前胚胎发育中异染色质重塑的规律。此外,该研究提出激活型DUX4对建立人ZGA阶段的抑制型组蛋白H3K9me3修饰具有重要贡献,为更好地理解生命之初转录因子与组蛋白修饰之间复杂的调控网络提供了新的见解。
本文的第一作者包括3344体育官方入口直博生徐睿敏,广东省第二人民医院李森博士,3344体育官方入口吴秋博士、李翀博士和广东省第二人民医院蒋满喜博士。高绍荣教授,欧湘红教授,刘晓雨教授与王晨飞教授是本文的共同通讯作者。该研究得到了科技部、国家自然科学基金委、广州生物岛实验室以及上海市科委等项目的支持。
参考文献
Becker, J.S., D. Nicetto, and K.S. Zaret, H3K9me3-Dependent Heterochromatin: Barrier to Cell Fate Changes. Trends Genet, 2016. 32(1): p. 29-41.
Nicetto, D., et al., H3K9me3-heterochromatin loss at protein-coding genes enables developmental lineage specification. Science, 2019. 363(6424): p. 294-297.
Matoba, S., et al., Embryonic development following somatic cell nuclear transfer impeded by persisting histone methylation. Cell, 2014. 159(4): p. 884-95.
Liu, W., et al., Identification of key factors conquering developmental arrest of somatic cell cloned embryos by combining embryo biopsy and single-cell sequencing. Cell Discov, 2016. 2: p. 16010.
Burton, A., et al., Heterochromatin establishment during early mammalian development is regulated by pericentromeric RNA and characterized by non-repressive H3K9me3. Nat Cell Biol, 2020. 22(7): p. 767-778.
Macfarlan, T.S., et al., Embryonic stem cell potency fluctuates with endogenous retrovirus activity. Nature, 2012. 487(7405): p. 57-63.
Whiddon, J.L., et al., Conservation and innovation in the DUX4-family gene network. Nat Genet, 2017. 49(6): p. 935-940.
Hendrickson, P.G., et al., Conserved roles of mouse DUX and human DUX4 in activating cleavage-stage genes and MERVL/HERVL retrotransposons. Nat Genet, 2017. 49(6): p. 925-934.
De Iaco, A., et al., DUX-family transcription factors regulate zygotic genome activation in placental mammals. Nat Genet, 2017. 49(6): p. 941-945.
Wang, C., et al., Reprogramming of H3K9me3-dependent heterochromatin during mammalian embryo development. Nat Cell Biol, 2018. 20(5): p. 620-631.
Skene, P.J. and S. Henikoff, An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife, 2017. 6.
Copyright© 2011-2015 3344体育官方入口 - 3344体育网平台
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


