Stem Cell Reports|高绍荣/张艳平/乐融融课题组揭示H3K9me3在胚胎干细胞向二细胞样状态细胞转变过程中的作用
2023年 1月 12 日,来自3344体育官方入口高绍荣教授领导的团队在Stem Cell Reports杂志上发表了题为“Remodeling of H3K9me3 during the pluripotent to totipotent-like state transition”的研究成果。
该研究工作利用ULI-NChIP-seq绘制了胚胎干细胞多能性状态与二细胞样状态转变中H3K9me3修饰的全基因组图谱,并结合转录组关联分析发现H3K9me3修饰主要在二细胞样状态细胞的LTR逆转座子区域被大量去除且伴随着大量重复元件的转录激活,但是启动子区域的H3K9me3修饰的减少并不会导致基因表达激活。另外还发现TSC(Trophoblast stem cells,TSCs)特异性增强子相关的H3K9me3的去除可能与二细胞样状态细胞分化潜能的扩展有密切联系。该研究工作对于进一步优化体外类全能干细胞的培养体系、理解胚胎发育和细胞命运决定提供了重要的理论依据,为全能性获得过程中H3K9me3修饰的作用研究提供了参考。

全能性是指单个细胞能独立发育成完整个体的能力,受精作用和核移植都可以使细胞获得全能性。小鼠的受精卵和 2-细胞卵裂球具有全能性(Lu F et al.,2015)。研究全能性的分子特征不仅可以解决发育生物学的基本问题,而且也可以为临床转化和应用提供理论依据。但是早期胚胎(主要是1细胞期和2细胞期)材料的匮乏阻碍了对全能性分子机制的研究。
从受精开始到胚胎植入前发育阶段,尤其是合子基因组激活(Zygotic genome activation, ZGA)发生前后,整个基因组表观修饰层面(比如基因启动子区的H3K4me3修饰和H3K27me3修饰,以及重复元件上的DNA甲基化修饰和H3K9me3修饰等)发生了比较广泛和剧烈的重塑(Xu, R et al,2021),广泛的表观遗传重塑对全能性的恢复和正常的胚胎发育起着至关重要的作用(Xu, Q et al., 2018)。而ZGA与表观遗传重塑和全能性的获得密切相关(Lu F et al.,2015)。在小鼠中,ZGA主要发生在二细胞胚胎阶段(Bouniol et al.,1995)。转录是最基本的调控细胞生物事件的层面,ZGA的发生又是新生命发生的第一个转录事件,若ZGA不能正常启动和精准调控则会导致胚胎发育异常和疾病的发生(Falco et al.,2007; Gabriels et al.,1999; Geng et al.,2012; Guo et al.,2019)。ZGA主要特征表现为大量的转座子重复元件(如MERVL)和 2-细胞胚胎特异性基因 (如DUX,Zscan4)发生转录 (Eckersley-Maslin et al., 2018)。
转座元件在人类和小鼠基因组中占据比较高的比例( Chénais, B et al., 2012 )。大量的转座子发生胚胎发育阶段性特征性转录,而且有特殊功能(Fadloun A et al., 2013) 。转座元件与基因转录调控关系甚为密切(Fueyo, R et al., 2022) ,转座元件表观遗传也参与了细胞状态的维持和细胞命运决定调控,其中H3K9me3修饰对于植入前胚胎中逆转座子的沉默起到了重要作用(Wang, C et al., 2018; Xu, R et al., 2021; Xu, R et al., 2022; Yu H et al., 2022)。
有报道称极少部分的胚胎干细胞能够短暂进入类似于2细胞期胚胎的状态(Macfarlan et al., 2012;Zalzman et al.,2010),这个亚群的胚胎干细胞称之为二细胞胚胎样细胞(2-Cell-like Cells,2C-like cells)。2C-like cells不仅显示出类似于二细胞胚胎的转录组特征,例如细胞胚胎特异性的基因(如Dux,Zscan4基因簇)和重复序列(如MERVL)高度上调(Macfarlan et al.,2012),而且还显示出类似于二细胞胚胎的染色质结构(Eckersley-Maslin et al.,2016; Ishiuchi et al.,2015)。同时这些2C-like cells还具有扩大的发育潜力,既能够形成胚胎组织,还能够形成胚外组织 (Macfarlan et al., 2012)。因此2C-like cells为理解全能性的分子基础和了解ZGA的机理提供了一个独特的体外模型。
组蛋白H3第9位赖氨酸三甲基化(Histone 3 lysine 9 trimethylation,H3K9me3)在细胞全能性获得和细胞命运转变过程中发挥了重要的调控作用(Becker et al., 2016; Nicetto et al., 2019)。然而H3K9me3修饰在胚胎干细胞向2C-like cells转变过程中的作用,以及H3K9me3修饰的重塑与细胞潜能拓展之间的关联机制等重要的科学问题仍存在很大的研究空白。
为了进一步探索H3K9me3在多能性干细胞向二细胞胚胎样细胞转变过程中的相关作用,高绍荣课题组绘制了二细胞胚胎样细胞中H3K9me3的全基因组图谱,并联合转录组进行关联性分析,详细比较和分析了谱系分化相关的H3K9me3修饰重塑在2细胞胚胎样细胞转变过程中的作用。
研究人员首先利用携带MERVL-tdTomato和pZscan4c-EGFP双荧光报告系统的胚胎干细胞系,发现多能性胚胎干细胞向二细胞胚胎样干细胞转变过程中,二细胞胚胎样细胞的核内H3K9me3修饰明显减弱,且H3K9甲基转移酶(SUV39H1/2)的表达水平有所降低。通过比较胚胎干细胞、二细胞胚胎样细胞和二细胞胚胎H3K9me3的全基因组图谱发现H3K9me3修饰在胚胎干细胞、二细胞胚胎样细胞和二细胞胚胎中具有相似的分布模式,但是二细胞胚胎样细胞富集程度高于二细胞胚胎,低于胚胎干细胞。启动子区域和LTR逆转座子区域存在不同的调控特征。相较于胚胎干细胞,中间态细胞和2C-like cells启动子区域主要发生的是H3K9me3修饰的去除。然而,在2-细胞样状态转变中,在启动子区域发生H3K9me3的去除并不伴随基因转录的激活,而发生转录激活的基因的启动子区域的H3K9me3信号在胚胎干细胞中却很低。这一结果表明启动子区域的H3K9me3修饰并不是2-细胞样状态转变中主要的基因表达调控机制。H3K9me3修饰主要富集在胚胎干细胞、中间态细胞和2C-like cells的LTR逆转座子区域。在LTR逆转座子区域,中间态细胞继承了胚胎干细胞H3K9me3修饰的分布特征,而在2C-like cells中H3K9me3修饰被大量去除且伴随着大量重复元件的转录激活。此外,位于上调基因附近区域的转座元件更倾向于发生转录的激活,这些区域可能作为替代的启动子或者增强子增加了其附近基因的转录活性。2C-like cells的发育潜能要高于中间态细胞和胚胎干细胞,同时具有向胚内和胚外谱系分化的能力。研究者接下来分析了谱系分化相关的H3K9me3修饰在2-细胞样转变过程中的重塑。结果发现基因启动子区域ESC特异性的H3K9me3修饰在2C-like转变过程中是逐渐去除的,2C-like cells细胞中ESC特异性的H3K9me3修饰峰的数目最少。胚胎干细胞中大部分TSC特异性增强子被H3K9me3修饰所标记,而这些区域(TSC特异性增强子和TSC特异性超级增强子以及TSC特异性LTR增强子)的H3K9me3修饰的去除主要发生在2C-like cells细胞中。而在中间态细胞中,TSC特异性增强子区域的H3K9me3修饰仍然高度富集。因此细胞特性相关的H3K9me3的去除可能与2C-like cells分化潜能的扩展有密切联系。
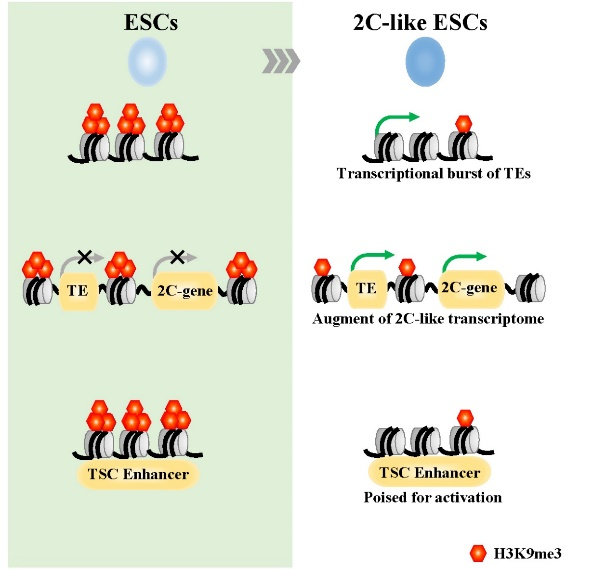
综上所述,这项研究提供了二细胞胚胎,二细胞胚胎样干细胞和胚胎干细胞之间表观遗传特性的详细比较和分析,探讨2-细胞样转变过程中H3K9me3修饰的重塑与细胞分化潜能拓展之间的关系,对于我们进一步理解ZGA、全能性和细胞命运转变的分子机制提供了帮助。
3344体育官方入口高绍荣课题组博士生李虎和博士生孙嘉彤为该论文的共同第一作者,高绍荣教授、张艳平副教授、乐融融教授为共同通讯作者。该研究得到了科技部重点研发计划、国家自然科学基金委、上海市科委等项目的支持。
文献引用
Lu, F., & Zhang, Y. (2015). Cell totipotency: molecular features, induction, and maintenance. National science review, 2(2), 217–225.
Xu, R., Li, C., Liu, X., & Gao, S. (2021). Insights into epigenetic patterns in mammalian early embryos. Protein & cell, 12(1), 7–28.
Xu, Q., & Xie, W. (2018). Epigenome in Early Mammalian Development: Inheritance, Reprogramming and Establishment. Trends in cell biology, 28(3), 237–253.
Bouniol, C., Nguyen, E., & Debey, P. (1995). Endogenous transcription occurs at the 1-cell stage in the mouse embryo. Experimental cell research, 218(1), 57–62.
Falco, G., Lee, S. L., Stanghellini, I., Bassey, U. C., Hamatani, T., & Ko, M. S. (2007). Zscan4: a novel gene expressed exclusively in late 2-cell embryos and embryonic stem cells. Developmental biology, 307(2), 539–550.
Gabriëls, J., Beckers, M. C., Ding, H., De Vriese, A., Plaisance, S., van der Maarel, S. M., Padberg, G. W., Frants, R. R., Hewitt, J. E., Collen, D., & Belayew, A. (1999). Nucleotide sequence of the partially deleted D4Z4 locus in a patient with FSHD identifies a putative gene within each 3.3 kb element. Gene, 236(1), 25–32.
Geng, L. N., Yao, Z., Snider, L., Fong, A. P., Cech, J. N., Young, J. M., van der Maarel, S. M., Ruzzo, W. L., Gentleman, R. C., Tawil, R., & Tapscott, S. J. (2012). DUX4 activates germline genes, retroelements, and immune mediators: implications for facioscapulohumeral dystrophy. Developmental cell, 22(1), 38–51.
Guo, M., Zhang, Y., Zhou, J., Bi, Y., Xu, J., Xu, C., Kou, X., Zhao, Y., Li, Y., Tu, Z., Liu, K., Lin, J., Yang, P., Gao, S., & Wang, Y. (2019). Precise temporal regulation of Dux is important for embryo development. Cell research, 29(11), 956–959.
Eckersley-Maslin, M. A., Alda-Catalinas, C., & Reik, W. (2018). Dynamics of the epigenetic landscape during the maternal-to-zygotic transition. Nature reviews. Molecular cell biology, 19(7), 436–450.
Chénais, B., Caruso, A., Hiard, S., & Casse, N. (2012). The impact of transposable elements on eukaryotic genomes: from genome size increase to genetic adaptation to stressful environments. Gene, 509(1), 7–15.
Fadloun, A., Le Gras, S., Jost, B., Ziegler-Birling, C., Takahashi, H., Gorab, E., Carninci, P., & Torres-Padilla, M. E. (2013). Chromatin signatures and retrotransposon profiling in mouse embryos reveal regulation of LINE-1 by RNA. Nature structural & molecular biology, 20(3), 332–338.
Fueyo, R., Judd, J., Feschotte, C., & Wysocka, J. (2022). Roles of transposable elements in the regulation of mammalian transcription. Nature reviews. Molecular cell biology, 23(7), 481–497.
Wang, C., Liu, X., Gao, Y., Yang, L., Li, C., Liu, W., Chen, C., Kou, X., Zhao, Y., Chen, J., Wang, Y., Le, R., Wang, H., Duan, T., Zhang, Y., & Gao, S. (2018). Reprogramming of H3K9me3-dependent heterochromatin during mammalian embryo development. Nature cell biology, 20(5), 620–631.
Xu, R., Li, C., Liu, X., & Gao, S. (2021). Insights into epigenetic patterns in mammalian early embryos. Protein & cell, 12(1), 7–28.
Xu, R., Li, S., Wu, Q., Li, C., Jiang, M., Guo, L., Chen, M., Yang, L., Dong, X., Wang, H., Wang, C., Liu, X., Ou, X., & Gao, S. (2022). Stage-specific H3K9me3 occupancy ensures retrotransposon silencing in human pre-implantation embryos. Cell stem cell, 29(7), 1051–1066.e8.
Yu, H., Chen, M., Hu, Y., Ou, S., Yu, X., Liang, S., Li, N., Yang, M., Kong, X., Sun, C., Jia, S., Zhang, Q., Liu, L., Hurst, L. D., Li, R., Wang, W., & Wang, J. (2022). Dynamic reprogramming of H3K9me3 at hominoid-specific retrotransposons during human preimplantation development. Cell stem cell, 29(7), 1031–1050.e12.
Macfarlan, T. S., Gifford, W. D., Driscoll, S., Lettieri, K., Rowe, H. M., Bonanomi, D., Firth, A., Singer, O., Trono, D., & Pfaff, S. L. (2012). Embryonic stem cell potency fluctuates with endogenous retrovirus activity. Nature, 487(7405), 57–63.
Zalzman, M., Falco, G., Sharova, L. V., Nishiyama, A., Thomas, M., Lee, S. L., Stagg, C. A., Hoang, H. G., Yang, H. T., Indig, F. E., Wersto, R. P., & Ko, M. S. (2010). Zscan4 regulates telomere elongation and genomic stability in ES cells. Nature, 464(7290), 858–863.
Eckersley-Maslin, M. A., Svensson, V., Krueger, C., Stubbs, T. M., Giehr, P., Krueger, F., Miragaia, R. J., Kyriakopoulos, C., Berrens, R. V., Milagre, I., Walter, J., Teichmann, S. A., & Reik, W. (2016). MERVL/Zscan4 Network Activation Results in Transient Genome-wide DNA Demethylation of mESCs. Cell reports, 17(1), 179–192.
Ishiuchi, T., Enriquez-Gasca, R., Mizutani, E., Bošković, A., Ziegler-Birling, C., Rodriguez-Terrones, D., Wakayama, T., Vaquerizas, J. M., & Torres-Padilla, M. E. (2015). Early embryonic-like cells are induced by downregulating replication-dependent chromatin assembly. Nature structural & molecular biology, 22(9), 662–671.
Becker, J. S., Nicetto, D., & Zaret, K. S. (2016). H3K9me3-Dependent Heterochromatin: Barrier to Cell Fate Changes. Trends in genetics : TIG, 32(1), 29–41.
Nicetto, D., & Zaret, K. S. (2019). Role of H3K9me3 heterochromatin in cell identity establishment and maintenance. Current opinion in genetics & development, 55, 1–10.
Copyright© 2011-2015 3344体育官方入口 - 3344体育网平台
地址:上海市四平路1239号 电话:021-65981041 传真:65981041


